在本文中,我们提出了一个增强到Apache SKAP,这是一个分布式计算框架,以适应高效的SQL查询基因组数据集。虽然可以使用现有技术导入基因组数据集作为SQL表,但这些技术对常见基因组查询的较差性能使它们成为下一代测序(NGS)数据的用户中的一个不吸引人的选择。
随着测序成本的下降,越来越多的研究中心投入大量的测序项目,目的是建立成千上万个基因组的庞大数据库及其相关的表型特征。例如,俄勒冈健康与科学大学(OHSU)和多发性骨髓瘤基金会(MMRF)分别对1000例急性髓系白血病(AML)和多发性骨髓瘤患者进行测序(1, 2)。在另一个例子中,国际癌症基因组联盟(ICGC)〔3〕正在对25000对肿瘤和正常样本进行测序,以编目50种不同癌症类型的遗传异常。每一个全基因组测序与Illumina的技术运行产生超过200 GB的数据。
获取这些数据,尽管对癌症治疗的进展至关重要,但对于研究人员和数据科学家来说仍然是一个挑战。
今天有两个层次的数据访问:一个顶部和底层。顶层涉及从存档中下载FASQ、BAM或VCF文件,如SRA〔4〕或CGHUB〔5〕,其中包含来自一个人或一个群体的排序的读取或变体。尽管这些档案利用了文件共享技术的技术来减少在因特网上的文件传输延迟——正如CGHUB使用GeEnTrOrn(6)的情况一样,需要传输的文件的大小使得下载速度变慢。例如,在100 Mbps因特网链路上下载具有60X覆盖全基因组测序(WGS)的250 GB BAM需要8小时。另一方面,底层涉及下载数据的子集的提取。用户要么从头开始开发软件来导航数据,要么使用shell脚本结合VCFoToS、SAMoTo工具和BEAToO刀的命令进行组合。这种做法在数据和用户之间增加了一层复杂性,原因有三:
1。必须创建脚本来分析这些实验
2。它要求用户在分布式环境中手动执行这些工具的执行,以适应日益增长的生成数据。
三。它通过创建用于转换文件的中间文件来创建存储开销。
假设基因组数据以万亿字节和字节字节的顺序驻留在分布式环境中(如7),我们建议更有效地替代两层数据访问是分布式数据检索引擎。顶部接入层上的这样的接口可以通过消除网络流量和在用户端进行二级处理的需要来提供按需数据。即使基因组数据库的拥有者不愿意提供这样的特征,数据检索接口仍然对底层用户的生产率有帮助。有了这样的接口,最终用户就不用担心脚本的方式来检索和比较来自不同来源的数据集(如原始读取、变体和注释数据)的数据。
在这项工作中,我们使用SPACK SQL,它是Apache Skine(8)框架的分布式SQL执行引擎。SCAPK是一种用户友好的高性能框架,它抽象了对象的分布式集合,它提供了大约80个操作符,它们可以通过OPER来映射这些对象,例如MAP、过滤器和GROMPBY,或者通过操作符对它们进行操作,例如,减少、计数和每一个。SARK在主从架构中组织一个集群,其中驱动程序(即,主)执行主程序,并将代码传递给工人(即奴隶),以执行它们所包含的分布式对象的那些部分。
数据模型
在这项工作中,我们假设所有的基因组数据是亚当格式。亚当(9, 10)是一个开源软件,它将基因组信息从其底层表示分离出来,目前被用作NIH BD2K翻译基因组学大数据中心的主要计算平台[7 ]。这样的分离消除了数据用户如何表示数据的负担。因此,亚当用户可以在不需要解析复杂文件的情况下对分布式存储进行操作,因为亚当支持并替换了目前由传统FASQ、BAM和VCF格式表示的所有级别的基因组数据。
亚当记录由以友好的基于列的格式存储的可序列化对象组成。它使用Apache AVro(11)作为数据序列化系统,它依赖于模式并将它们与数据一起存储。使用Apache Pald[ 12 ]系统存储序列化数据,该系统是基于谷歌Delmi耳(13)的柱状存储系统。实木地板通过对记录序列进行分组来创建存储块,并依次存储每个块的所有列。最后,假定实木地板提供了对Hadoop文件系统(HDFS)的写入和读取的内置支持,亚当透明地支持在HDFS上构建的分布式环境。
SpultSQL完全识别实木拼盘文件,并因此将亚当文件作为关系表,也推断出它们的模式。这允许用户从Sql SQL本地查询亚当文件。
问题
虽然SCAPK SQL提供了令人印象深刻的表达能力,因此它可以执行任何基因组查询,但是它的查询基因组数据的主要障碍是它在两个最经常遇到的查询上的慢性能:1)随机范围选择,和2)与间隔键连接。在一个对齐的读取集合中,随机范围的选择花费了几分钟的时间在一个小的集群中运行,这是非常困难的,因为SAMDoTS只需要几秒钟。幸运的是,我们使用的开源库(特别是亚当文件所依赖的PoPoice和其API Spice SQL用于它们的过滤)的快速演进改进了这些查询的执行顺序,如我们在结果部分中所示。关于两个表之间的间隔连接的执行,StaskSQL在其交叉产品上使用了明显的过滤执行。然而,考虑到基因组数据所涉及的大小,这样的方法是不现实的。例如,如果我们考虑10亿个对齐的读取与100万个变体之间的间隔连接,它们之间的交叉积是1015个记录,并且计算是非常缓慢的。
本文的贡献解决了第二个性能瓶颈:加入区间密钥。我们提出了一个修改SCAPK SQL,提高了间隔连接的效率,从而使它适合查询基因组数据。为此,我们使用间隔树来间隔在分布式设置中加入两个表。
相关工作
访问基因组数据的第一代工具涉及诸如SAMOToS[14 ]、VCFToo[[ 15 ] ]、BAMToo[ 16 ]和BEDoToo[ 17 ]之类的包。虽然功能强大,但这些工具需要广泛的编程技术来打开和解析不同格式的文件、分配缓冲区和操作各种字段。此外,考虑到这些工具对单节点计算性能进行优化,用户需要在分布式环境中手动并行化它们。
第二代相关软件涉及基因组查询语言(GQL)(18),它通过SQL类接口提供基因组数据收集的干净抽象。然而,GQL的支持仅限于对整个山姆规范的一小部分字段的查询,并且还需要额外的人工努力来支持分布式环境。
第三代利用Hadoop生态系统来轻松地提供分布式环境下的数据需求。例如,基因计量查询语言(GMQL)〔19〕使用Apache PHOG,它是抽象地图缩减操作的高级语言,以支持元数据管理和变量和注释数据之间的查询。在另一个例子中,NeXBIO(20)使用HASBASE(Hadoop的NoSQL关键值存储)来支持类似性质的数据。然而,这些工具的范围不包括FASQ或BAM格式中的原始数据。
实施
本节介绍了如何修改SCASTSQL以增加对基于范围的连接的支持。修改的第一步涉及SCAPQSQL的语法,我们扩展了语法以简化这些查询的语法。接下来,在描述我们对SARK SQL执行引擎的修改之前,我们提供了该修改使用的间隔树和间隔林数据结构的简要描述。
句法
虽然SCALL SQL的现有语法足以满足用户在间隔重叠条件下描述两个表之间的连接,但对于常规使用该操作的基因组集合的用户来说,它看起来复杂和违反直觉。如果我们考虑表A(ASTAR:LUN,AUD:LUN,ACHR:String)和B(BSTAR:Load,Detri:Burn:BCHR:String),那么根据String SQL的当前语法,一个间隔连接看起来如下:
SELECT ∗ FROM A JOIN B ON ( aChr = bChr AND a S ta r t < aEnd AND b S ta r t < bEnd AND ( aStart < bStart AND aEnd > bStart OR bStart < aStart AND bEnd > aStart ) )
为了消除这种频繁操作的复杂性,我们用两个额外的关键字,即RangeJoin和Geang-Meple来增强SARKSQL的词汇。在基于间隔的连接的情况下,前者关键字替换联接,后者以两个元组为参数,指定重叠条件,并且是ON条件的唯一参数。
使用这些新关键字,可以将前面示例的查询键入如下:
SELECT ∗ FROM A RANGEJOIN B ON GENOMEOVERLAP( ( aS tar t , aEnd , aChr ) , ( bS ta r t , bEnd , bChr ) )
区间树
连接评估中最昂贵的部分涉及搜索两个区间数组之间的重叠。我们的实现利用区间树数据结构,它是从O(n log n)时间中的N个区间的集合构建的二叉树,并且需要O(log n)时间来找到其集合的哪个间隔与给定的查询间隔重叠。注意,同一操作的SurvivSQL使用最新的强制执行需要二次时间。
在这一点上,我们提醒感兴趣的读者如何构建和搜索区间树。
树的每个节点包含两个对象。第一个是键,它是存储在子树中的间隔集合的中间点,该子树是根植在节点上的。第二个对象是包含与键重叠的那些间隔的重叠列表。例如,考虑图1的存储树的间隔树。
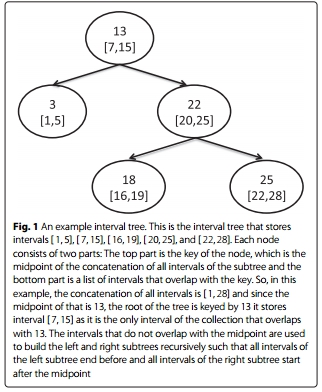
区间〔1, 5〕、〔7, 15〕、〔16, 19〕、〔20, 25〕和〔22, 28〕。根的关键是13,因为这是所有区间的结合的中点,即[1, 28 ]。此键仅与间隔[7, 15 ]重叠,这是根的重叠列表的唯一内容。
节点的子树存储那些不与其键重叠的间隔。左子树包含所有端点小于密钥的间隔;对称地,右子树包含所有起始点大于密钥的间隔。
为了搜索特定间隔是否与间隔树的任何间隔重叠,一个扫描根的重叠列表以搜索可能与查询重叠的间隔,并根据查询间隔的相关位置继续遍历树。d遇到的节点的密钥。当输入间隔在节点的键之前结束时,搜索仅继续到左子树。分别在查询间隔开始大于密钥时,搜索仅继续向右。在输入间隔和密钥之间的重叠的情况下,搜索继续到两个子树。假设使用图1的区间树搜索具有[17, 23 ]的所有重叠区间。从根开始,在对根的重叠列表进行快速扫描后,返回空集,根和间隔之间的比较表明搜索应该继续向右子树,该根子树植根于以22为关键字的节点。节点的重叠列表的快速扫描检测到[20, 25 ]是解决方案的一部分,并且由于查询间隔与节点的键重叠,所以搜索继续向左和右子树。以同样的方式进行,查询结果返回一组区间[20, 25 ]、[16, 19 ]和[22, 28 ]。