目录
面向真实科学问题的Java课程设计
1. 项目简介
2. 实现方法与细节
Class UnitSeq
Method Summary
Class Unit
Method Summary
Class CodeList<T extends java.lang.Number>
Method Summary
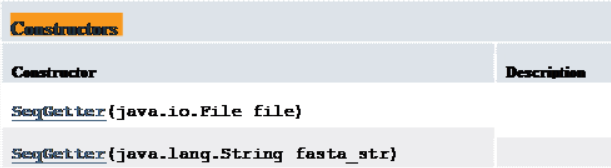
Class SeqGetter
Constructor Summary
Method Summary
Class SeqMan
Constructor Summary
Class SeqLib
Constructor Summary
Class SeqLibNotEncodedException
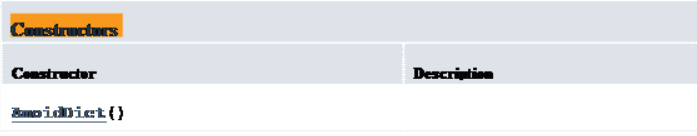
Class AmoidDict<T>
Constructor Summary
Class KmerBasedDict
Constructor Summary
Class EncoderHandler
Method Summary
Enum EncoderType
Class ResetHandler
Method Summary
Class OneHotEncodingHandler
Method Summary
Class BagOfWordsHandler
Method Summary
Class TFIDFHandler
Method Summary
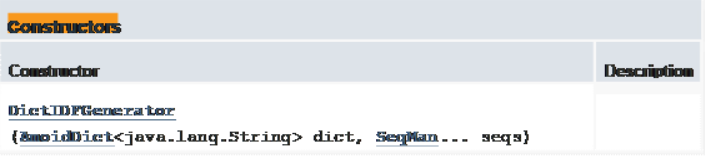
Class DictIDFGenerator
3. 实验结果与分析
1. 开发环境
2. 开发平台
3. 项目内容
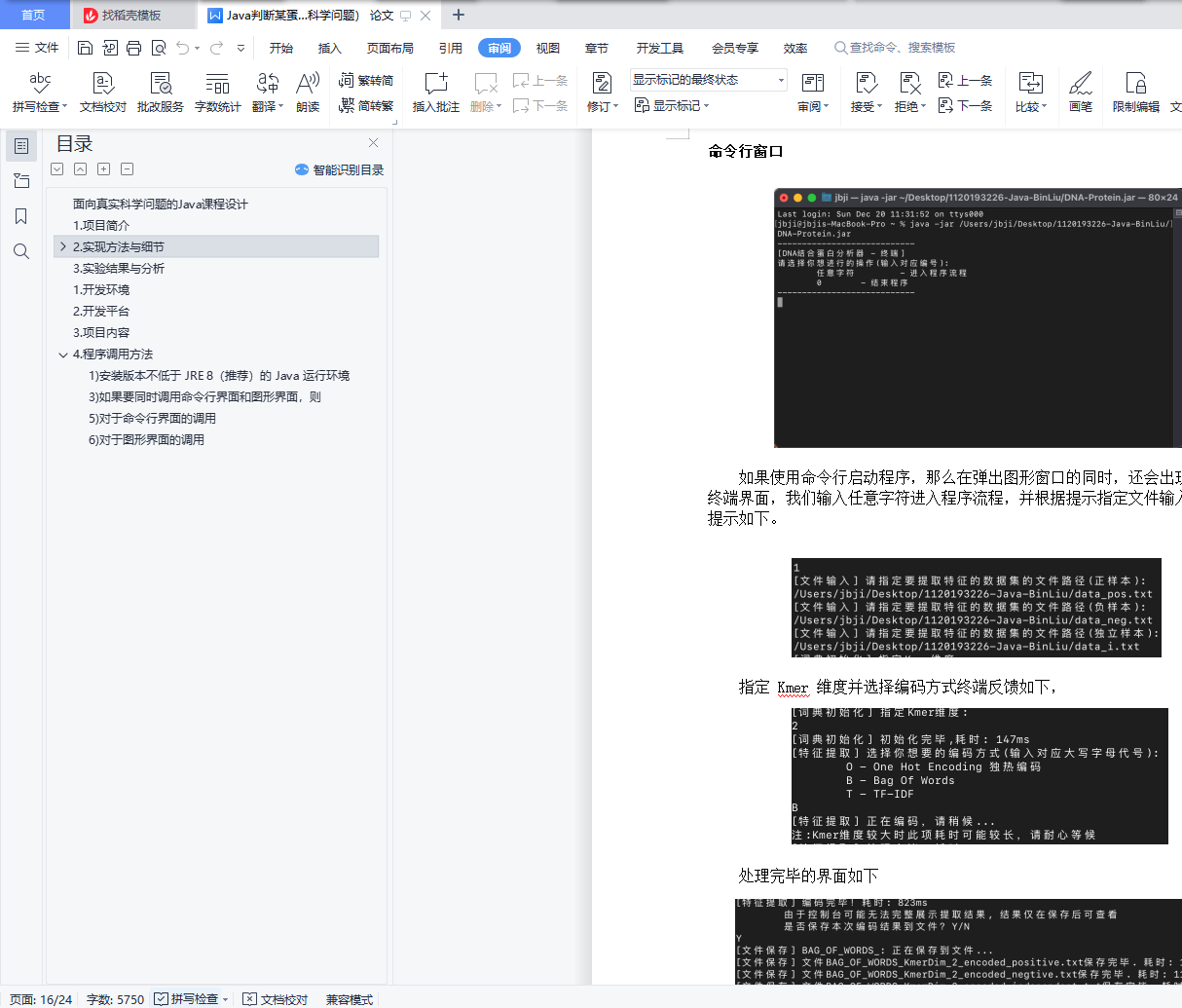
4. 程序调用方法
1) 安装版本不低于 JRE 8(推荐)的 Java 运行环境
3) 如果要同时调用命令行界面和图形界面,则
5) 对于命令行界面的调用
6) 对于图形界面的调用
1. 项目简介
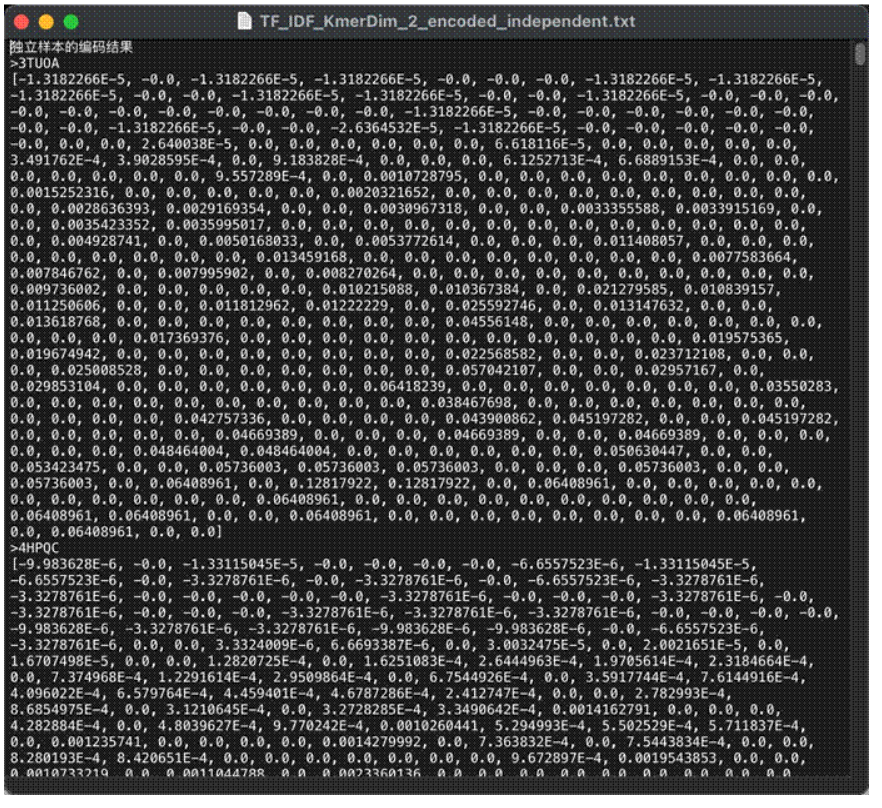
简单地说,本项目要实现的最终目标是,判断某蛋白质序列是否是DNA结合蛋白。报告给出的具体实现已经实现的结果是,将给定的蛋白质序列FASTA 格式(序列名/注释+单个字母表示的核酸或氨基酸)输入,转换成一串便于分类器理解的向量。向量间数学特征的差异,可以反映不同序列输入的文字特征和语义特征,有助于区分不同的蛋白质序列。
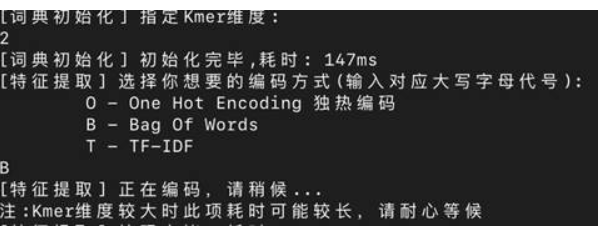
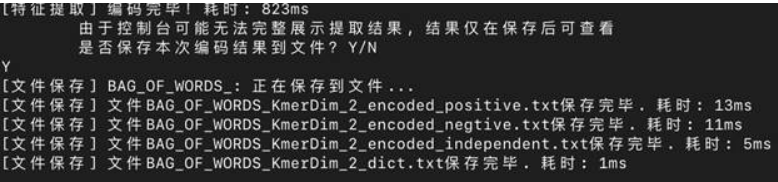
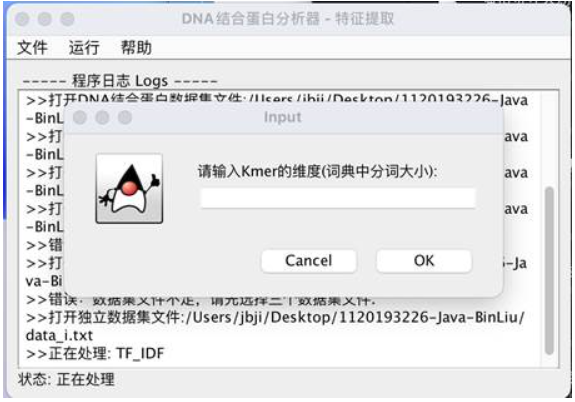
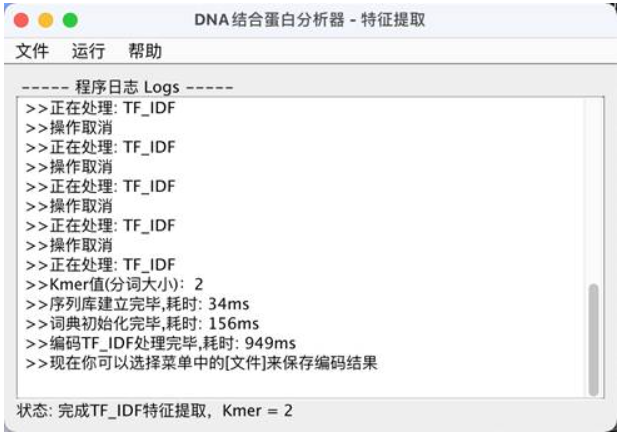
项目初步实现的功能包括,运用Kmer分词方法创建词典,给出了包括One Hot Encoding 、 Bag Of Words 、 TF- IDF在内的三种特征提取方法。用户可以通过有限的图形交互界面来实现基于文件输入的交互,并获得向量序列的文件输出。
项目简单实现了数据与表现的分离,提供了一定的可扩展性,既可以通过调用项目实现的有限内容来获得向量序列的文件输出,也可以通过调用项目包含的底层数据逻辑来构造进一步的机器学习模型。